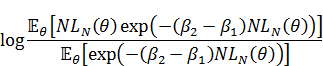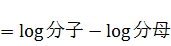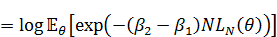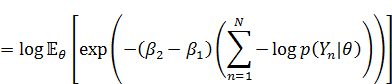概要
2017年8月9日に国立がん研究センターは、がん治療拠点の約半数にあたる全国188の病院について、癌患者の5年後の生存率データを初めて公表しました(毎日新聞の記事)。報告書は国立がん研究センターが運営するウェブサイトからダウンロードできます(ここ)。報告書をダウンロードしようとすると注意点を記したポップアップが表示されます。大切な部分を抜粋すると以下です。
本報告書には、施設別の生存率を表示していますが、進行がんの多い少ない、高齢者の多い少ないなど、施設毎に治療している患者さんの構成が異なります。そのため、単純に生存率を比較して、その施設の治療成績の良し悪しを論ずることはできません。
一般に高齢者が多い病院ほど、進行癌(ステージが進んだ癌)が多い病院ほど、その病院の生存率は下がるわけです。それならば、統計モデリングで年齢と進行度(ステージ)の影響を取り除いて(専門的な言葉で言えば「調整して」)病院の良し悪しを論じてみようというのがこの記事の内容になります。しかし、あくまでも「手法」の節で書いた仮定のもとでの推定結果であり、真実として断定するものではありませんのでその点はご理解ください。
なお、影響を取り除く前の(カプランマイヤー法で算出した)実測生存率のヒストグラムは以下になります。病院によってかなりばらつきがありそうに見えます。何もしないで比べると「国立研究開発法人国立がん研究センター中央病院」や「がん研有明病院」など大きな病院の生存率が高いです。患者の平均年齢と平均的な進行度が低いためと思われます。

結果
それでは結果から先に述べます。
以下では病院ごとの生存率や手術率を比較するために、癌種t・病院hにおける男性比率を0.5・平均年齢を60・平均進行度を2.5(おおよそステージIIに相当)に仮に固定して議論をすすめます。
=== 2018.2.6 追記 ===
この部分が分かりにくかったようなので補足します。今知りたいことは「あなたが60歳でステージIIだったらどこへ行くのが5年生存率が高いか」です。しかし、病院によっては若い人が多かったり、重症な人が多かったりして、そのままの生存率を単純に比較できません。そこで、統計モデリングによってすべての病院の平均年齢と平均進行度を仮想的に揃えることで、病院にだけ依存する生存率の差が残るわけです。ここでは一例として平均年齢を60・平均進行度を2.5に固定して算出しています。とにかく揃えればよいので、仮に平均年齢を50・平均進行度を1.5に固定してもよいです(この場合、生存率は全体的に高い方へ少しずれますがランキングは変わりません)。
癌種ごとの生存率のヒストグラム
生存率の推定値(MCMCサンプルの中央値)のヒストグラムは以下になります。

驚くべきことに胃・大腸・肺においてはほとんど病院の差がありません。がん診療連携拠点病院、さすがです。肝癌と乳癌は少し病院によって差がありますので、それぞれbest10を紹介します。ただし、病院による差は提供されたデータ以外の要因(他の病気をもつ患者が多い少ないなど)も含まれます。検討によってそういう情報が重要と分かれば、今後データとして収集して公表する必要があると思います。
肝癌における生存率best10の病院

黒点は(MCMCサンプルの)中央値、横に伸びる線は80%ベイズ信頼区間です。中央値の高い順に並び替えています。ベイズ信頼区間を見ると分かる通り、病院による差はそこまで明らかではありません。
「大垣市民病院」があまりに良いので病院のコメントを読むと「4.肝(C22)は切除症例に限る。」と書いてありました。これだと切除していない患者さんとその死亡数が考慮されていないので、フェアではなく正しいランクとは言えません。ただし、他の癌種における結果を見ると、全てを考慮してもかなり良い生存率である可能性はあります。
仮に「大垣市民病院」を除くと「信州大学医学部附属病院」が1位となります。
乳癌における生存率best10の病院

グラフの見方は肝癌の場合と同じです。
1位は「愛知県がんセンター中央病院」です。圧倒的な手術率もさることながら、手術以外の影響(病院による効果)も1位です。謎の民間療法に頼らず、こういうところに入院したいものです。
=== 2018.12.8 追記 ===
識者の方から、がんセンターは一般的に合併症のないピュアな癌患者を受け入れる傾向が強いので生存率が高いのでは、とコメントをいただきました。合併症のデータも重要で調整する必要があるかもしれません。今後の課題と思います。
生存率への年齢・進行度・手術率の影響(オッズ比)

点は中央値、横に伸びる線は80%ベイズ信頼区間です。例えば、胃癌の年齢_10のOdds ratioの中央値は約0.5ですが、これは「ある病院における患者の平均年齢が10上がると『生存率/死亡率』が約0.5倍になる」ことを意味します。性別_0.1は男性比率が0.1上がることを意味します。他も同様です。
結果を見ると、予想通り性別の影響はあまりなくて年齢や進行度の影響が強いです。特に胃癌においては年齢の影響が強く、乳癌においては進行度の影響が強いことが示唆されています。
手術率への年齢・進行度の影響(オッズ比)

グラフの見方は上の場合と同様です。肝癌において進行度の影響が低そうなのが意外に思いました。このあたり、病院によって生存率がばらついていることの原因があるのかもしれません。
その他
ここでは記しませんが、他にも色々知ることができました。例を挙げます。
- 手術の好きな病院・嫌いな病院
- 手術率が比較的低いわりに生存率がまあまあ高い病院
- 生存率worst10の病院
手法
データの抽出
このような報告書を公表するのは英断であり、 “がん診療連携拠点病院ががん患者さんの治療に透明性を確保し、拠点病院全体として責任をもって取り組んでいる意気込み” (報告書からの抜粋)をまさに感じることができました(なかには難癖つけて公表を拒否している病院があり最後にまとめました)。一点、ちょっと残念なのは報告書がpdf(580ページほど)であり、利活用が大変しにくいことでした。今回は素敵なRによるスクレイピング入門を出している株式会社ホクソエムの市川さんと牧山代表取締役に担当してもらい、Rのtabulizerパッケージを使って抽出しました。
データの難所
この報告書には188の病院が含まれていて、各病院について5つの癌(胃、大腸、肝、肺、女性乳房)のデータが含まれています。1つの病院の1つの癌のデータ例を以下に示します(数値は架空のものです)。

個人情報保護の観点から、「1人以上10人以下」の場合に-(ハイフン)に置き換えられています。これがこのデータの解析の難易度を大幅に引き上げています。
ハイフンへの対策は以下のようにしました。
- 合計から数値が計算できる場合は計算して置き換える。
- 例: 「観血的治療の実施」(=外科的手術の実施)の有と無の合計が「対象数」(=患者数)となるため、上記の例では「観血的治療の実施_無」は100-91=9と算出できる。
- 例: 「(100.0 - 生存状況把握割合(%))*対象数」を四捨五入して「打ち切り数」が算出できる。
- 場合の数を列挙し、統計モデリングに組み込む。
- 例: 年代のところでは、「50歳未満」と「80歳以上」の合計が18人と分かります。あり得る組み合わせは(10,8), (9,9), (10,8)の3通りだけです。この各場合の確率を算出して合計して尤度とします(StanとRでベイズ統計モデリング (Wonderful R)の7.8節と11章、ベイズモデリングの世界の第II部の講義3を参照)。
生存率はどの値を使うか
打ち切りを考慮してカプランマイヤー法で算出した生存率を使う方法がまず考えられますが、対象数の大小に由来する生存率の推定幅をうまく組み込むのが容易ではありません。また、がん診療連携拠点病院の意気込みが凄くて、現時点で生存状況把握割合は95%以上が多く、今後は100%に近づきそうです。これらを踏まえて、対象数と5年後生存数を用いた二項ロジスティック回帰にしました。*1
対象数が少ない場合への対応
元の報告書では、対象数が50人未満の場合、定された生存率の信頼性が低くなるため公表しないとしてハイフンになっています。本解析においても対象数が少ない場合、年齢や進行度のデータがほぼすべてハイフンとなって、列挙する場合の数が非常に多くなります。そこに推定の時間を取られるのは本意ではないため、同様に50人未満の場合は解析対象から除外しました。
統計モデル
使用したモデルは以下になります。














青色の項は女性乳房以外の癌において存在し、赤色の項は肺癌のみに存在します。inv_logitはロジスティック関数です。ここでデータは以下です。
: 癌種のインデックス(tissue)。
が癌種の数で、ここでは
。
: 病院のインデックス(hospital)。
が病院の数で、ここでは報告書にならって患者数が50以上のみを解析対象としたため、
。
: 生存数
: 対象数(患者数)
: 観血的治療の実施有の数(外科的手術をした人数)
: 各年代に属する人数
: 各進行度に属する人数
: 性別が男性の人数
: 肺癌において、部位が小細胞である人数
: 平均
の正規分布を0.5, 0.6, 0.7, 0.8で切って5つの領域の面積をそれぞれ
に割り当てます。その閾値です。
: 平均
の正規分布を0.2, 0.3, 0.4で切って4つの領域の面積をそれぞれ
に割り当てます。その閾値です。
ちなみに、発見経緯のデータは進行度に反映されると仮定して今回は使用していません。
推定するパラメータは以下です。推定にはStanとRを用いました。
: 生存率
: 手術率
: 男性比率
: 肺癌における小細胞率
: 癌
, 病院
における「平均年齢」。*2
: 癌
, 病院
における「平均進行度」。*3
: 各年代の比率
: 各進行度の比率
: 生存率に対する説明変数以外の影響。病院差。
: 手術率に対する説明変数以外の影響。病院差。
,
: 回帰係数
: 生存率の病院差の標準偏差。データから推定します(階層モデル)。
: 手術率の病院差の標準偏差。データから推定します(階層モデル)。
年齢と癌の進行度の影響を取り除くため、これらを説明変数とした二項ロジスティック回帰にするのがポイントです。さらに手術率も年齢や進行度の影響をうけると考えられるので上記のモデルとなっています。と
がそれぞれ別の多変量正規分布から生成されるモデルも試してみましたが、現段階では少しデータが足りないようで、MCMCが収束しませんでした。また探索的な解析からは、年齢や進行度によって生存率や手術率が指数関数的に落ちるのではなく線形に近いことが示唆されたため、今回は非線形な関数を含まないモデルとしました。実際のコードは上記の数式に加えてハイフンの処理が入るのでかなり複雑になります。今回はコードの解説を省略します。
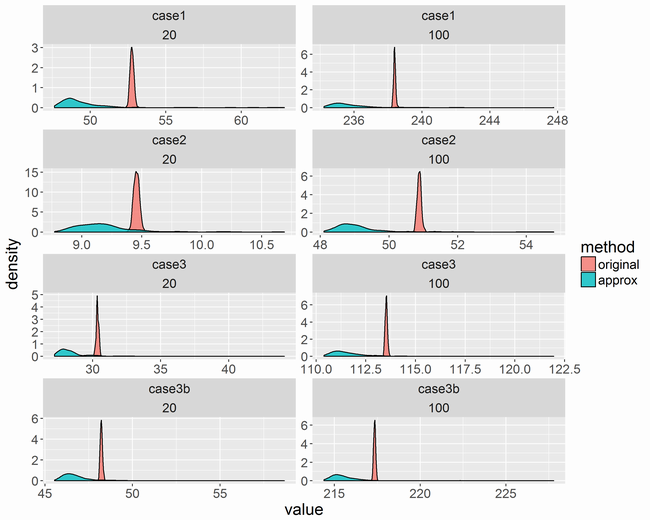
最後にモデルの事後予測チェックの図を以下に載せておきます。生存数の実測値と予測値(点は中央値、縦に伸びる線は80%ベイズ信頼区間)は以下です。

生存率のカプランマイヤー法による実測値と予測値(点は中央値、縦に伸びる線は80%ベイズ信頼区間)は以下です。肝癌は少しあてはまりが悪いですが、全体的に問題なく推定できていると思いました。

手術件数の実測値と予測値(点は中央値、縦に伸びる線は80%ベイズ信頼区間)は以下です。

その他にも癌, 病院
における「平均年齢」と「平均進行度」の推定などがうまくいっているかもチェック済みです。
後記
謝辞
がん診療連携拠点病院と国立がん研究センターがん対策情報センターがん登録センター院内がん登録室に感謝します。相談に乗ってくれた@Med_KUさんと@happyningenさんに感謝します。
展望
全ての癌種の最新かつ時系列のデータで再解析がしたいです。もし患者レベルのデータが利用できれば、さらに良い解析ができます。いつか解析できることを楽しみにしています。
難癖つけて公表しなかった残念な病院一覧
治療のレベルはお察し。毎回更新したいです。
- 王子総合病院
- 岩手県立中部病院
- 岩手県立胆沢病院
- 秋田県厚生農業協同組合連合会秋田厚生医療センター
- 秋田県厚生連 由利組合総合病院
- 山形市立病院済生館
- 公立富岡総合病院
- 埼玉医科大学国際医療センター
- 国保直営総合病院君津中央病院
- 都立駒込病院
- 帝京大学医学部附属病院
- 杏林大学医学部付属病院
- 福井大学医学部附属病院
- 国立病院機構岩国医療センター
- 山口県厚生農業協同組合連合会周東総合病院
*1:打ち切りがある場合は5年後生存数を正確に得ることはできませんが、生存状況把握割合が非常に高いので、打ち切りに含まれる生存数を「打ち切り数*既知の生存数/対象数」で近似的に算出しました。
*2:本来は患者の年齢と生存か否かが結び付けれればさらに良い解析ができますが、現段階の情報ではこうやって多少強引に平均年齢を算出する他なさそうです。年代の人数は割と山型の分布なので、これで良さそうです。
*3:年代と同様です。しかし、癌種によっては「ステージIとIVは多いけどIIとIIIは少ない」ということがあり、進行度を要約するために平均+正規分布を使うのではなく、他の指標と分布を使った方がよいかもしれません。検討の余地があります。