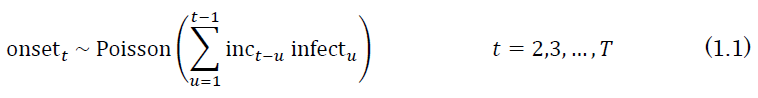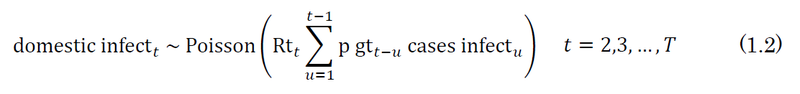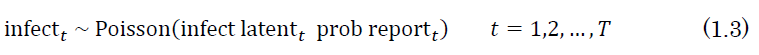先日の西浦先生のニコ生の発表を聞いていない人はぜひ聞いてください。
モデルとデータを以下のリポジトリでオープンにしていただいたので、モデルについて僕が分かる範囲内で少し解説を加えたいと思います。
github.com
実効再生産数を推定するコードが2種類ありまして、最尤推定(Maximum Likelihood Estimation, MLE)を使ったMLE版(Sungmok Jungさん作成)と
、ベイズ推定版(Andrei Akhmetzhanovさん作成)があります。どちらもコンセプトはほぼ同じで、実装が若干異なります。この記事では、ベイズ推定版(以降、元コードと呼びます)の流れを簡単に説明し、その後でその拡張を試みます。
大きく分けて「データの集計」「back projection」「実効再生産数の推定」の3つの部分からなります。
データの集計
まずは日付ごとの人数のデータを作ります。
- 発病日(onset)があるデータ(12526人)については発病日を使います。日付ごとに人数の合計を算出します。
- その他のデータ(2872人)については、診断日または報告日(report)を報告日と呼んで、日付ごとに人数の合計を算出します。
なお、発病日と報告日を両方持っている人は(悲しいことに)存在しません。
back projection
再生産方程式は、「ある時刻において感染者は何倍に増えたか?」を記述する式なので、「感染者の人数」と「感染から感染までの時間(生成時間, generation time)」をベースに考えます。そこで、上記の「発病日の人数」や「報告日の人数」を「感染日の人数」に変換します。この変換をback projectionと呼んでいます。
具体的には以下の式に従うと考えて、ある日 における感染者数
における感染者数  を推定します。
を推定します。
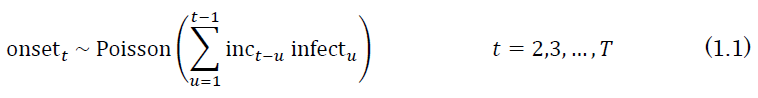
ここで、 はある日
はある日 における発病者数です。
における発病者数です。 は潜伏期間(incubation period, 感染から発病までの期間)の分布です。発病者数と潜伏期間の分布はデータとして与えます。
は潜伏期間(incubation period, 感染から発病までの期間)の分布です。発病者数と潜伏期間の分布はデータとして与えます。
例えば、 の発病者数
の発病者数  の平均は、1日目における感染者数
の平均は、1日目における感染者数  にその感染者たちが9日後に発病する確率をかけたもの、2日目における感染者数
にその感染者たちが9日後に発病する確率をかけたもの、2日目における感染者数  にその感染者たちが8日後に発病する確率をかけたもの、…、9日目における感染者数
にその感染者たちが8日後に発病する確率をかけたもの、…、9日目における感染者数  にその感染者たちが1日後に発病する確率をかけたもの、の総和で定まるだろうということを表しています。この(合計が10になるように)掛け算して総和をとる部分は畳み込み(convolution)と呼ばれ、本記事でたびたび登場します。
にその感染者たちが1日後に発病する確率をかけたもの、の総和で定まるだろうということを表しています。この(合計が10になるように)掛け算して総和をとる部分は畳み込み(convolution)と呼ばれ、本記事でたびたび登場します。
元コードではback projectionを実効再生産数の推定の前に行います。そこで使用している{surveillance}パッケージとその推定方法については、Med_KUさんの以下のブログ記事が詳しいのでぜひそちらを参照してください。
mikuhatsune.hatenadiary.com
英語になりますが、こちらにも詳しい説明があります。
元コードでは海外帰国と国内の感染者に分けて、それぞれに対してback projectionを行っています。まず2872人の報告日のデータについて、発病日→報告日の分布を与えて、発病日を計算するback projectionを行っています。すると、12526+2872人分の発病日のデータとなります。さらに発病日のデータについて、潜伏期間の分布を与えて、感染日を計算するback projectionを行っています。
back projectionではスムージングもあわせて実行されます。スムージングは「本来の感染者数は日付についてそこまでギザギザになっておらず、ある程度なめらかでしょう」という仮定を反映しています。スムージングの強さはハイパーパラメータとして与えます。
また、このback projectionの結果、求まる感染者数は小数の値になります。さらに変換前後で合計人数を変えないように、感染者数の合計=変換前の人数の合計 が成り立つように正規化しています。
実効再生産数の推定
元コードではback projectionを最尤推定で求めた感染者数を使って、実効再生産数Rtを推定しています。
まずは簡単のため、「感染しているけど、発病日あるいは報告日のデータとしてまだ上がってきていない」という「報告遅れ」を考慮しないモデルを考えます。具体的には、以下の再生産のモデル式に従うと考えて、実効再生産数を推定します。
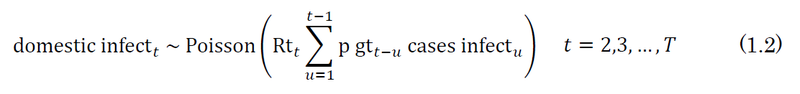
ここで、 はある日
はある日  における国内の感染者数です。
における国内の感染者数です。 はある日
はある日  における海外帰国の感染者数と国内の感染者数の合計です。
における海外帰国の感染者数と国内の感染者数の合計です。 は生成時間の分布です。感染者数と生成時間の分布はデータとして与えます。
は生成時間の分布です。感染者数と生成時間の分布はデータとして与えます。
ポアソン分布の平均が、実効再生産数と「感染者数と生成時間の分布の畳み込み」の積になっている点に注目です。ある日  の実効再生産数が1程度であることは、「その時点より過去の感染者数」と「生成時間の分布」から計算される「現在の感染者数」が同程度で、感染が広がっていない(狭まってもいない)ことを表します。
の実効再生産数が1程度であることは、「その時点より過去の感染者数」と「生成時間の分布」から計算される「現在の感染者数」が同程度で、感染が広がっていない(狭まってもいない)ことを表します。
左辺が小数の値をとるので厳密にはポアソン分布ではないのですが、階乗をガンマ関数で表現することでポアソン分布と同じ形の尤度を考えることができます。*1
西浦先生が説明されていましたが、無症状者の割合を時刻によらず一定と仮定すれば、無症状者まで含めた感染者数に対して、上記の再生産のモデル式がそのまま成り立ちます。よって、推定した実効再生産数をもとにした議論はそのまま適用可能です。
Stanによる実装
元コードにおいてモデルはStanで実装されています。元コードでは以下の部分が少し難解です。
- 畳み込みに使う分布をStanコード内で定義している。
- 連続値を許すポアソン分布の代わりに、rateパラメータが1のガンマ分布を用いている。
- 尤度がガンマ分布の二度漬けになっている。
- 報告遅れを平均ベースで考慮している。
そこで、ここでは元コードの意図と推定結果をほとんど変えないままで、少しシンプルにしたStanコードを紹介します。
- 2~10行目: すべての日(ただし初日は除く)における、
xとyの畳み込みを求める関数です。計算をラクにするため、あらかじめyを逆順に並べたものを引数として渡します。
- 12~16行目: 連続値を許すポアソン分布の定義です。
lamがゼロを含む場合にはもう少し定義を頑張る必要がありますが、ここではこれで大丈夫です。正規化定数はclosed formで表現できないのですが、1に近いのでこれで近似する意味です(参考URL1, 参考URL2)。
- 23行目:
p_gt_revは生成時間の分布の値(逆順)です。Stan内で計算するよりRで計算して渡す方がラクなのでそうしています。
- 35行目: (1.2)式に相当する部分です。
.*はvector型どうしの要素ごとの積です。
推定時間はiter=10000で、私のX1 carbonで1chainあたりおよそ20秒でした。
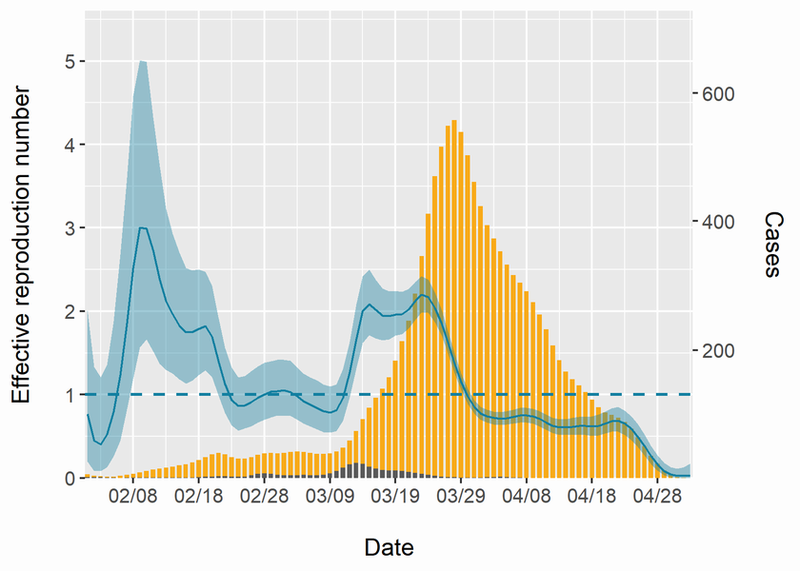
back projectionして求めた感染者数は元コードと同じものを使い、上記のStanコードで推定した結果の図が以下になります。実線と灰帯はそれぞれ実効再生産数の中央値と95%信用区間(左軸)、棒グラフはback projectionで求めた感染者数(右軸)です。
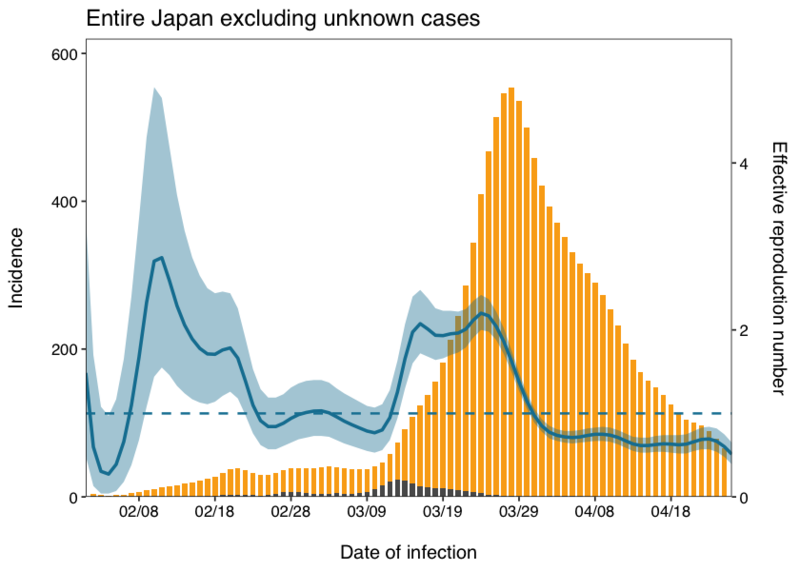
以下の図はMLE版の推定結果です。インターネット上でよく議論されていた図です。この図と大体同じ結果が得られていると分かります(上の図では横軸を05/03まで伸ばしている点は異なりますが)。
モデルの拡張
西浦先生の話と質疑応答にもありましたが、以下のような拡張は考えうると思います。
- 検査のバイアス・陽性率の変動は考慮していない → 今のところ難しい。他のデータが必要そう。
- 生成時間の分布をあらかじめ与えている → 分布の算出に使用したデータが少ないので検討の余地あり
この記事ではひとまず上記の拡張はさておき、元コードの意図を極力保持しながら、仮定を緩和するような拡張方法を考えます。具体的には、「back projectionのモデル化」と「報告遅れのモデル化」を行いましたのでそれらを説明します。
back projectionのモデル化
元コードにおける上記の流れの懸念点として、back projectionで確定的に求めた感染者数を、実効再生産数の推定においてデータとして与えてしまっていることが挙げられます。これをやると、back projection自体の不確定性を考慮していないことに相当し、信頼区間を過小評価することにつながります。
そこで、この節ではback projectionも実効再生産数のモデルに組み込みます。上記で説明したback projectionの(1.1)式をモデルに組み込んだだけで、残りの部分は元コードに忠実です。Stanコードは以下になります。
- 26行目:
p_inc_revは潜伏期間の分布の値(逆順)です。Rコード参照。
- 27行目:
p_rep_revは感染から報告までの時間の分布の値(逆順)です。Rコード参照。
- 32行目: 感染者数に対して1階差分のスムージングをかけています。
s_smoothはそのばらつきの大きさでデータから推定されます。
- 33~36, 40~43行目: back projectionの変換前後で合計人数が変わらないようにするために、要素の合計が変換前の合計人数と一致するようなパラメータを定義しています。
simplexにスケーリングを掛けることで実現するテクニックを使っています。国内domesticと海外帰国imported、発病onsetと報告report、の合計4種類のback projectionがあることに注意。
- 51~55行目:
simplexの空間でスムージングしています。スムージングのばらつきs_smoothを共有しています。
- 57~61行目: back projectionの(1.1)式に相当する部分です。
上記をコンパイルするときに、「~の左辺がパラメータの非線形変換を含んでいたらヤコビアンを調整してね」という旨のお知らせが表示されますが、ここでは変換は定数をかけたり足したりする線形変換なので、ヤコビアンの調整は不要と思います。
推定を実行するRコードも載せておきます。推定時間はiter=6000で、私のX1 carbonで1chainあたりおよそ12分でした。
- 10~34行目: データの集計部分です。
- 37~59行目: 生成時間、潜伏期間、感染から報告までの時間のそれぞれ分布を定義しています。本来は連続値をとる確率分布を、日付ごとの離散的な分布に直すため、累積分布関数から計算している点に注意してください。特に最後の、感染から報告までの時間の分布は、潜伏期間+発病から報告までの時間 という二つの確率変数の和の分布になるので、ここでも分布の畳み込みが出てきます。互いに独立な確率変数の和の累積分布関数については、例えばこの講義ノートを参照してください。
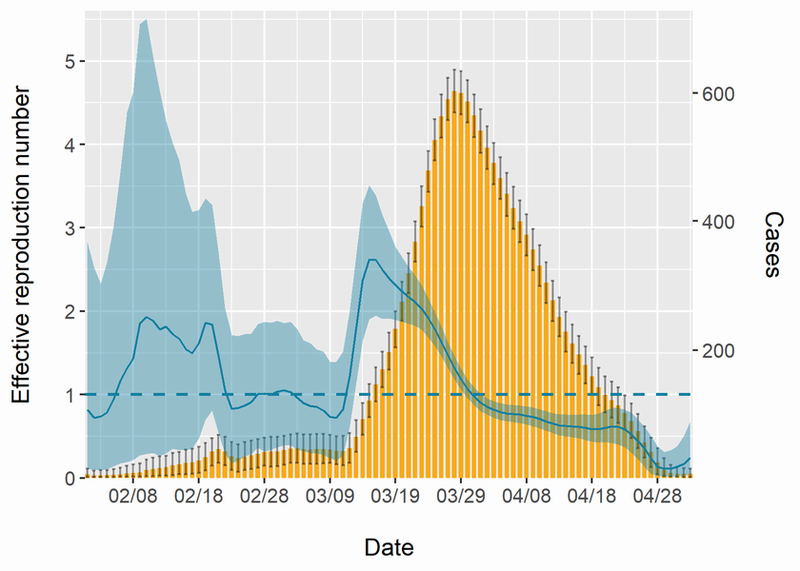
結果は以下の図です。棒グラフは推定された感染者数の中央値、エラーバーは95%信用区間です。先ほどと比べて、実効再生産数の信頼区間が全体的に少し広くなっていることが分かります。
報告遅れのモデル化
元コードでは、「back projectionで求めた感染者数」を「感染から報告までの時間の累積分布関数の値」で割ることで、まだどのデータにも上がってきていない感染者数の分を増やして実効再生産数の推定に用いていました。その方法では、人数の多寡にかかわらず常に確定的に水増しされることになり、少人数の場合の不確定性をやはり考慮できていません。結果として、まだどのデータにも上がってきていない人数が含まれるであろう、ここ2週間ぐらいの実効再生産数の信頼区間が過小評価されることにつながります。
そこで、この節では報告の遅れも実効再生産数のモデルに組み込みます。ただし、元コードと同じ方法ではなく、以下の式に従うと仮定しました。
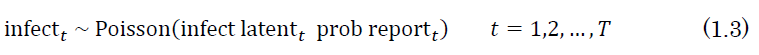
ここで、 はback projectionで求めた、ある日
はback projectionで求めた、ある日  における感染者数です。
における感染者数です。 は報告が遅れている人まで含めた、
は報告が遅れている人まで含めた、 における感染者数です。
における感染者数です。 は
は  においてどれぐらいの人数がデータとして上がってきているかを表す確率です。
においてどれぐらいの人数がデータとして上がってきているかを表す確率です。
Stanコードは以下になります。
- 28行目: 感染から報告までの時間の累積分布関数の値です。
- 38~41, 53~55行目: 報告が遅れている人も含めた感染者数を定義しています。
- 71~73行目: (1.3)式に相当する部分です。
Rコードの説明は省略します。推定時間はiter=6000で、私のX1 carbonで1chainあたりおよそ30分でした。
結果は以下の図です。棒グラフは報告が遅れている人まで含めた感染者数を表します。先ほどと比べて、ここ2週間ぐらいの信頼区間が少し広くなっていることが分かります。
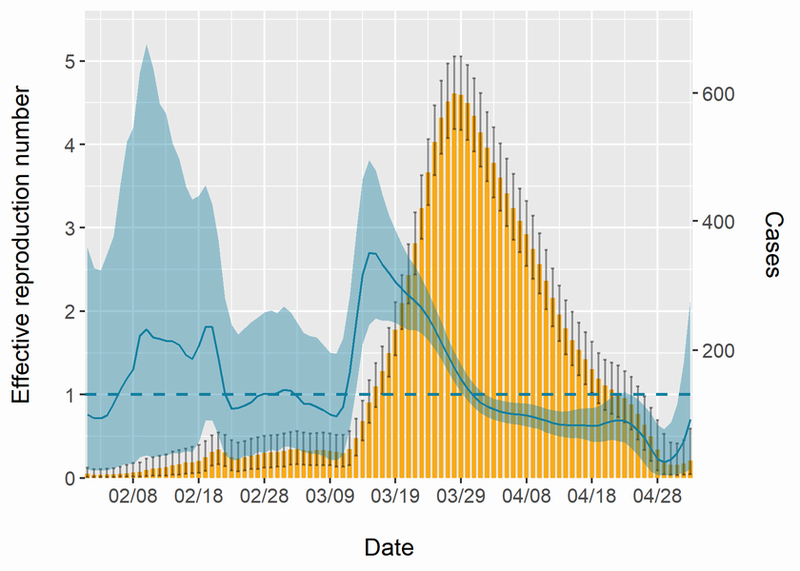
ここ2週間の不確定性を含めた推定結果が重要だと思うので、個人的には最後のモデルを推したいです。
enjoy!!